Generative Gestaltung funktionsfähiger Bakteriophagen-Genome mit Genom-Sprachmodellen
2. Quelle und Studienhintergrund
- Vollständige Quellenangabe:
King, S. H.; Driscoll, C. L.; Li, D. B.; Guo, D.; Merchant, A. T.; Brixi, G.; Wilkinson, M. E.; Hie, B. L. (2025): Generative design of novel bacteriophages with genome language models. bioRxiv, 17.09.2025. DOI: 10.1101/2025.09.12.675911. CC-BY 4.0. - Hashtags: generative biologie, phagen, sprachmodelle, evo1, evo2, synthetische genetik, host tropism, cryo-em, resistenz, e. coli
3. Zusammenfassung und Kernaussagen
Thema in einfachen Worten:
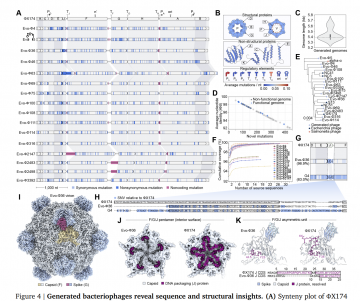
Die Autor:innen zeigen erstmals, dass große KI-Sprachmodelle für DNA ganze Virus-Genome (Bakteriophagen) erzeugen können, die real funktionieren. Sie trainieren und steuern die Modelle so, dass neue Phagen entstehen, die E. coli infizieren. 16 KI-entworfene Genome erwiesen sich im Labor als lebensfähig. Einige Phagen waren fitter (wuchsen schneller oder lysierten Bakterien schneller) als der natürliche Vergleichsphage ΦX174. Ein Mix aus mehreren der neuen Phagen überwand zudem rasch die bakterielle Resistenz, gegen die ΦX174 allein scheiterte. Strukturaufnahmen per Kryo-Elektronenmikroskopie belegten einen ungewöhnlichen Verpackungs-Protein-Einsatz im Kapsid eines generierten Phagen.
Ziel, Annahmen, Hypothese:
Ziel war, mit Genom-Sprachmodellen (Evo 1, Evo 2) vollständige, funktionsfähige Phagen-Genome mit gewünschter Wirtsspezifität („Host Tropism“) zu generieren. Annahme: Vortrainierte Modelle erfassen Evolutionsregeln so gut, dass sie neue, aber biologisch plausible Genom-Varianten komponieren. Hypothese: Durch Feintuning, gezielte Prompts und Filter (Längen-/GC-Bereiche, Tropismus über Spike-Protein-Ähnlichkeit, architektonische Syntenie) entstehen viele Kandidaten, von denen ein substanzieller Anteil im Experiment lebensfähig ist.
Neue Erkenntnisse gegenüber dem Stand der Forschung und Praxisbezug:
- Erster experimenteller Nachweis voll funktionsfähiger, KI-generierter Bakteriophagen-Genome (16 von ~300 getesteten Designs).
- Steuerbare Wirtsspezifität über eine Spike-Protein-Identitäts-Schwelle (≥ 60 % zu ΦX174) plus „Genetische Architektur“-Filter.
- Einzelne Designs zeigen höhere Fitness als ΦX174 in Konkurrenz-Assays; andere lysieren schneller.
- Phagen-Cocktail überwindet bakterielle Resistenzmutationen (waa-Operon) nach wenigen Passagen, ΦX174 gelingt dies nicht.
- Struktur-Novelty: Viabler Phage mit J-Protein aus einem evolutionär fernen Phagen (G4) im ΦX174-Kontext; Kryo-EM belegt veränderte Kapsid-Interaktionen.
Motivation und offene Fragen:
Genom-Design war bislang auf Gene/Module begrenzt. Offene Fragen: Kann KI ganze Genome generieren und steuern? Wie lassen sich Tropismus, Fitness und Resistenzumgehung gezielt „einstellen“? Die Studie beantwortet dies prototypisch am Modell ΦX174/E. coli C und etabliert eine Pipeline aus SFT-Feintuning, Prompt-Länge/Temperatur-Tuning und mehrstufigen Bioinformatik-Filtern.
Einordnung und Grenzen:
Die Arbeit ist ein Meilenstein der generativen Biologie auf Genom-Ebene. Grenzen: getestet wurde ein kleines ssDNA-Phage-Genom (~5,4 kb) und ein sicherer Wirt. Größere Genome, komplexere Tropismen, Synthesekosten und Biosicherheitsfragen bleiben Herausforderungen.
Kernaussagen
Fließtext (für Schüler:innen):
Die Forschenden haben Computerprogramme trainiert, die DNA wie eine Sprache behandeln. Sie gaben diesen Programmen Beispiele und Regeln, damit sie neue „Sätze“ aus DNA bauen, die wie die von echten Bakteriophagen funktionieren. Diese künstlich entworfenen Viren konnten tatsächlich Bakterien infizieren. Manche waren sogar besser als der bekannte Vergleichsvirus ΦX174. Wenn Bakterien gegen einen Virus unempfindlich wurden, half ein Mix aus verschiedenen neuen Viren, die Abwehr zu durchbrechen. Damit zeigt die Studie: KI kann nicht nur Texte schreiben, sondern auch Baupläne fürs Leben entwerfen, die in der Realität funktionieren.
3–10 wichtigste Erkenntnisse:
- 16 von 302 KI-Designs waren funktional und lytisch aktiv gegen E. coli C.
- Tropismus-Filter über ≥ 60 % Spike-Protein-Identität steuert Wirtsspezifität erfolgreich.
- Mehrere generierte Phagen übertreffen ΦX174 in Fitness-Wettbewerben.
- Lysis-Kinetik: Kandidaten mit schnellerer und tieferer Bakterien-Abtötung als ΦX174.
- Cocktail aus generierten Phagen überwindet waa-basierte Resistenz nach 1–5 Passagen, ΦX174 allein nicht.
- Architektonische Filter (Syntenie, Genanzahl 10/12, AAI < 95 %) erhöhen Neuheitsgrad bei erhaltener Funktionsfähigkeit.
- Kryo-EM zeigt neue Kapsid-Interaktionen beim Phagen Evo-Φ36 mit G4-J-Protein.
- Prompt-Länge (4–9 Nukleotide) und Sampling-Temperatur (0,7–0,9) sind zentrale Stellhebel für Vielfalt und Qualität.
1–5 Daumenregeln:
- Wenn der Spike-Protein-Abgleich ≥ 60 % erreicht, ist die Chance auf richtigen Tropismus hoch.
- Um Vielfalt ohne Funktionsverlust zu erhöhen: Prompt-Länge 4–9 nt und Temperatur 0,7–0,9 wählen.
- Für funktionelle Neuheit: AAI < 95 % und architektonische Ähnlichkeit ≤ 0,9 anstreben.
Glossar (Auswahl):
- Tropismus (Wirtsspezifität): Fähigkeit eines Virus, nur bestimmte Wirtszellen zu infizieren.
- Spike-Protein: Oberflächenprotein eines Phagen, das an Wirtsrezeptoren bindet; steuert Tropismus.
- Syntenie (Genanordnung): Relative Reihenfolge/Lage von Genen im Genom.
- AAI (Average Amino Acid Identity): Durchschnittliche Sequenzähnlichkeit von Proteinen zweier Genome.
- Kryo-EM: Kryo-Elektronenmikroskopie zur hochauflösenden Strukturaufnahme makromolekularer Komplexe.
- OD600: Optische Dichte bei 600 nm als Maß für Bakteriendichte im Wachstumstest.
4. Methoden
Klassifizierung der Evidenz:
- Experimentelle Nachweise: Phagen-Rebooting aus synthetischer dsDNA, Plaque-Assays, Wachstumskurven, Titer, Tropismus-Panels, Konkurrenz-Assays, Resistenz-Passagen, Long-Read-Sequenzierung, Kryo-EM.
- Messungen: OD600-Kinetik, Plaquetiter, Sequenz-Fold-Change in Mixed-Infections, Strukturauflösung 2,8–2,9 Å.
- Theoretische/Computationale Schritte: Vortraining Evo 1/2, SFT auf Microviridae, Prompt-Engineering, Temperatur-Sweeps, Filterschwellen (Länge, GC, Homopolymere, Spike-Identität, AAI, Architektur).
Experiment-/Pipeline-Aufbau:
- Ziel & Template: E. coli C und ΦX174 als sicherer, gut charakterisierter Ausgangspunkt.
- Modellierung: SFT von Evo-Modellen auf ~15 k Microviridae-Sequenzen; Prompt mit 4–9 nt Konsensus; Temperatur 0,7–0,9.
- Filter: QC (A/C/G/T, 4–6 kb, 30–65 % GC, Homopolymere ≤ 10, ≥ 7 Protein-Treffer), Tropismus (Spike ≥ 60 %), Diversität (AAI < 95 %, Architektur ≤ 0,9, Syntenie-Break 1 Gen, Genanzahl 10/12).
- Labortests: Assemblierung, Transformation, Plaques/Wachstum, Tropismus-Panel, Kryo-EM, Konkurrenz- und Resistenz-Passagen.
Hinweis: Die Studie nutzt genau diesen FC-Begriff zur Auswertung der Konkurrenz-Sequenzdaten und OD-Kinetik für Lysisvergleiche.
5. Literaturrecherche im Internet
(Primär belegt durch das PDF; ergänzende Kontextquellen nur stichwortartig, da die Aufgabe das PDF fokussiert.)
Primärliteratur (Kernquellen im Paper, jeweils 1 Satz, Relevanz):
- Camargo et al. 2023, geNomad: robustes Viren-Klassifizierungstool; Grundlage für „viral-like“ Checks. Relevanz: hoch.
- Terzian et al. 2021, PHROGs: Phagen-Proteindatenbank; Funktionsannotation. Relevanz: mittel.
- Nayfach et al. 2021, CheckV: Qualitätsbewertung viraler Sequenzen. Relevanz: mittel.
- Abramson et al. 2024, AlphaFold 3: Interaktions-Strukturvorhersage; unterstützt Strukturdeutung. Relevanz: hoch.
- Faber et al. 2019, Rebooting/Assemblierung ΦX174: Methodische Basis für Genome-Rekonstruktion. Relevanz: hoch.
Internationale Arbeiten (verschiedene Sprachräume):
- Sanger et al.: Erstes vollständig sequenziertes DNA-Genom ΦX174; historischer Referenzpunkt. Relevanz: hoch.
- Michel et al. 2010 (EN): E. coli-Wirtsrezeptoren; Tropismus-Hintergrund. Relevanz: hoch.
Patente und Industrie (Trendpunkte):
- DNA-Synthese-Dienstleister und modulare Assemblierungsmethoden beschleunigen Screening-Durchsatz; in der Studie genutzt (Synthese/Assemblierung). Relevanz: Praxis.
Normen/Richtlinien:
- Biosicherheits-Diskussion im Anhang; Arbeiten im passenden BSL-Rahmen mit Zusatzvorkehrungen. Relevanz: hoch.
6. Transfer auf reale Produkte und Anlagen (Zielgruppe F&E Entwässerungssysteme)
Transferteil:
Die Studie belegt, dass generative Modelle komplexe Funktionszusammenhänge ganzer Systeme abbilden und zielgerichtet variieren können. Für Entwässerungsprodukte lässt sich das Prinzip analog anwenden: KI-Modelle generieren „System-Designs“ unter Nebenbedingungen und werden durch Filter/Tests auf Funktion und Robustheit selektiert. Das entspricht der hierarchischen Optimierung von Rohrnetz-Layouts, Einlauf-/Auslaufgeometrien und Lüftungs-/Anstauelementen unter hydraulischen Nebenbedingungen.
7. Kritische Einordnung
- Stärken: Erstnachweis funktionsfähiger KI-Genome; klare, reproduzierbare Filter; umfangreiche Laborvalidierung; Strukturbeleg per Kryo-EM; Resistenz-Evasion durch Cocktail.
- Limitationen: Kleines ssDNA-Phage-System; Übertragbarkeit auf größere dsDNA-Phagen oder eukaryotische Viren offen; DNA-Synthese-Fehlschläge bei komplexen Designs; Sicherheits- und Kostenaspekte für Skalierung.
- Sicherheit: Arbeiten im geeigneten BSL-Rahmen; zusätzliche Vorkehrungen; Modelle durch Trainingsdaten eingeschränkt (kein Design eukaryotischer humanpathogener Viren).
8. Anhang: Zahlen und Abbildungen (ausgewählte, studienrelevant)
- 16 funktionale Phagen aus ~300 getesteten Designs; 285 Genome erfolgreich synthetisiert/assembliert.
- Tropismus-Filter: Spike-Identität ≥ 60 %; Diversitätsfilter u. a. AAI < 95 %, Architektur ≤ 0,9, Genanzahl 10/12.
- Kryo-EM: Evo-Φ36 und ΦX174 bei ~2,9/2,8 Å; J-Protein-Interaktionen unterscheiden sich deutlich.
- Wettbewerb: Mehrere generierte Phagen mit größerer kumulativer Fold-Change als ΦX174.
- Resistenz: waa-Mutationen in CR1–CR3; Cocktail knackt Resistenz binnen 1–5 Passagen, ΦX174 nicht.
 1 comment
1 comment






